
PROJET 3BIM

PROJET 3BIM




Ce site constitue le support de présentation de notre projet 3BIM, réalisé dans le cadre de notre troisième année à l’INSA de Lyon au département Biosciences - Bioinformatique et Modélisation.
Aujourd’hui, pour connaître l'avancée de la recherche et les innovations dans un domaine particulier, nous pouvons étudier l’attribution des prix Nobel. Cependant, les prix Nobel sont souvent accordés de nombreuses années après la découverte. Le temps de latence est trop élevé, ils ne sont donc pas un bon caractère pour déterminer l'innovation. Une autre idée serait de regarder dans la base de données des brevets mais la recherche ne conduit pas nécessairement à un intérêt industriel et donc à un dépôt de brevets. Enfin, il est possible d’être alerté des sujets biologiques en vogue en s’informant sur d'autres prix attribués aux chercheurs chaque année, mais cela restreindrait trop notre recherche.
Plusieurs objectifs nous ont motivés dans la réalisation de ce projet.
Dans un premier temps, notre formation amènera beaucoup d’entre nous à travailler dans le domaine de la recherche. Chaque projet implique une étape de recherche bibliographique, qui peut parfois être assez périlleuse. Certaines pistes de recherches sont par exemple très peu documentées, il nous est donc apparu utile de disposer d’un outil capable d’orienter nos recherches documentaires.
Notre premier objectif était donc de développer un outil d’aide à la documentation, permettant de visualiser le développement d’une thématique de recherche en lien avec la biologie. Cet outil permettrait ainsi de voir l’émergence de nouveaux thèmes mais également leur disparition.
Nous avons fait le choix de réduire notre étude au domaine de la biologie pour concorder à notre formation liée à la bioinformatique, mais également pour réduire notre domaine de recherche.
L’utilité finale de notre outil serait de caractériser l’innovation dans les thématiques biologiques, quantifier les chances de développement d’une thématique particulière et donc prédire lesquelles ont le plus de chance d’être les prochaines tendances.
Nous avons principalement utilisé le langage Python. En effet, Python est largement utilisé en bioinformatique, il dispose de nombreuses bibliothèques disponibles dédiées à l'étude informatique de données biologiques.
Nous avons également utilisé le langage R pour présenter nos résultats. Il nous a également permis de réaliser des tests statistiques dont il dispose.
Nous avons décidé d'utiliser la base de données PubMed pour l'extraction de nos données. PubMed est le principal moteur de recherche de données bibliographiques dans les domaines de la biologie et de la médecine. C’est un moteur gratuit donnant accès à la base de donnée MEDLINE rassemblant des citations et des résumés d’articles de recherches biomédicales. On compte ainsi un grand nombre de données disponibles. On retrouve plus de 24 millions de citations datant de 1950 à 2014 issues de 5000 revues biomédicales disponibles gratuitement. C’est donc le moteur de recherche le plus pertinent pour connaître l’état de l’avancée des recherches dans le domaine de la biologie.
Pour extraire les données disponibles sur PubMed, nous avons utilisé la librairie Biopython développée par OBF (Open Bioinformatics Foundation), un groupe de volontaire qui favorise le développement d’outils bioinformatiques dans la recherche biologique. En particulier, nous avons exploité le package Entrez de Biopython qui permet de récupérer des informations sur les articles tels que l’identifiant, le titre, la date de publication, l’abstract ou encore les identifiants des articles par lequel il est cité. Les informations récoltées se présentent sous la forme de containers usuels du langage Python (dictionnaires, listes, ...).
Un problème que nous avons rencontré lors de l’extraction des données, était que seulement certains articles disposaient des mots clés associés. Pour les autres articles, il nous a fallu générer des mots clés par nous-même. Pour ce faire, nous avons utilisé un traitement de texte permettant d’extraire des mots importants à partir d’un paragraphe. Nous avons appliqué cet algorithme sur le titre et les abstracts des articles sans mots clés associés. L’algorithme fait ressortir les mots qui se retrouvent fréquemment en excluant les mots simples (comme "the" ou "of"). Les limites de cette méthode sont que les mots clés générés ne sont pas toujours pertinents, ou assez souvent moins pertinents que les mots clés proposés par Pubmed. De plus, il peut arriver que le thème principal ne soit pas abordé dans l’article.
L'idée de notre projet se base sur l'article Dynamics on expanding spaces: modeling the emergence of novelties. Cet article explique comment on peut modéliser l’apparition d’innovations. L’innovation désigne quelque chose qui possède un caractère de nouveauté. Les innovations se retrouvent dans des domaines variés comme les sciences exactes, humaines, l'industrie, l'art, le social...
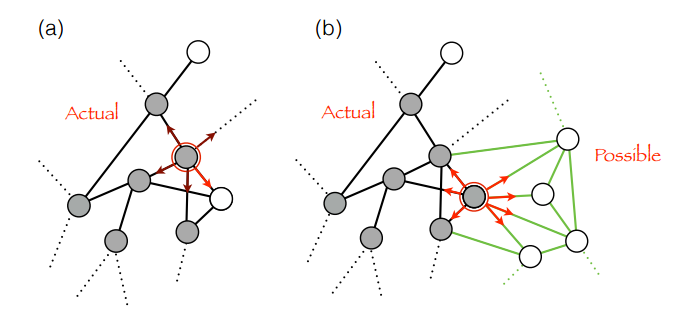
Une notion importante est alors le concept d’adjacent possible. Ce concept a été introduit dans les travaux de Stuart Kaufman. L’adjacent possible correspond ici aux boules blanches qui représentent des idées non découvertes à un instant donné. Ces idées sont "adjacentes" car elles se situent à un pas d’être découvertes par rapport aux idées déjà connues.
Se déplacer sur une boule blanche dans le graphe (c'est-à-dire découvrir une nouvelle idée) fera apparaître de nouvelles boules blanches liées à la première et donc élargir le graphe. Cela permet de schématiser le phénomène d’apparition d’innovations.
Dans l'article, l'apparition d'innovation est modélisée par une version modifiée de l'urne de Polya. On se place dans une urne qui contient plusieurs boules de couleur. Les boules peuvent représenter des thèmes, des idées...
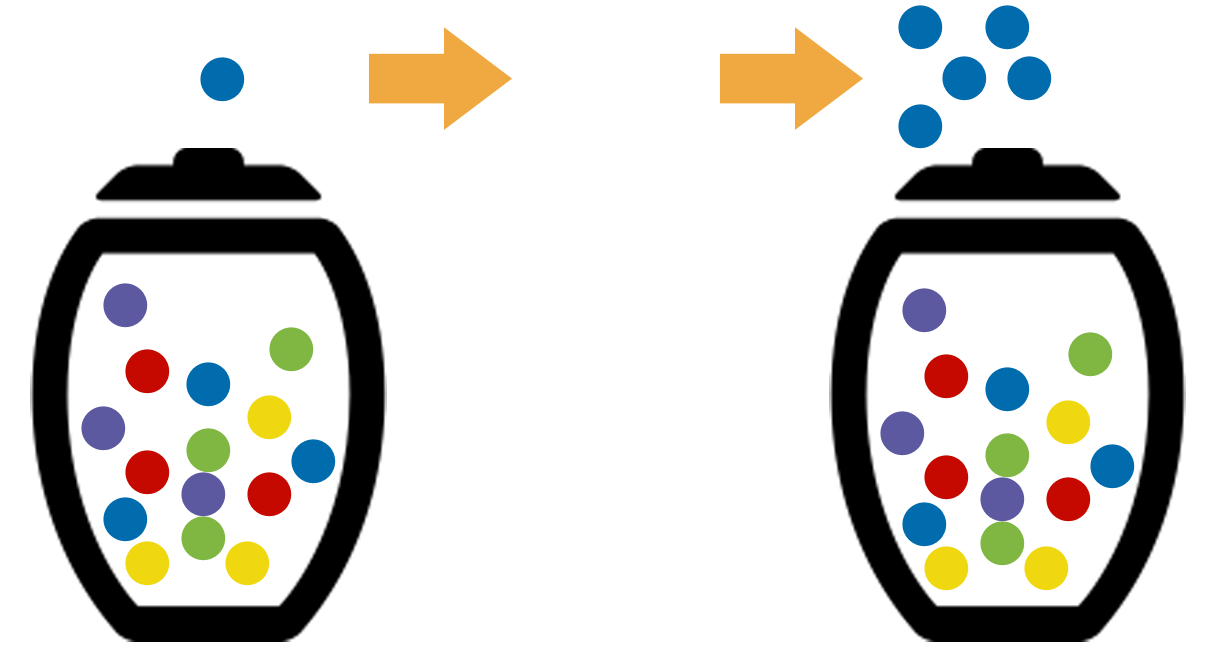
Dans l'article, l'apparition d'innovation est modélisée par une version modifiée de l'urne de Polya. On se place dans une urne qui contient plusieurs boules de couleur. Les boules peuvent représenter des thèmes, des idées... À l'état initial, un certain nombre de boules de différentes couleurs est présent dans l'urne. En parallèle, on considère une liste (S) contenant des couleurs (vide au départ).
À chaque fois qu'une boule d'une couleur présente dans la liste est tirée, des boules de la même couleur sont ajoutées dans l'urne. La probabilité de piocher cette couleur est alors augmentée pour le prochain tirage. On peut alors parler d’effet de "buzz" ou un effet de "rich get richer" qui est retrouvé dans un urne de Polya classique.
À chaque fois qu'une boule d'une couleur non présente dans la liste est tirée, des boules de nouvelles couleurs sont ajoutées dans l'urne. La couleur est aussi ajoutée dans la liste S. Les boules de nouvelles couleurs correspondent à des nouveaux concepts et donc à de l'innovation. Cela se base sur le fait qu'une innovation peut engendrer d'autres innovations. Dans ce modèle, la croissance des nouvelles couleurs est logarithmique.
À long terme, en quantifiant le nombre de boules par couleur, les données obéissent à une loi de Zipf. Cette loi modélise initialement la fréquence des mots dans un texte. La fréquence d'occurrence f(n) d'un mot est liée à son rang n dans l'ordre des fréquences par une loi de la forme f(n) = K/n où K est une constante.
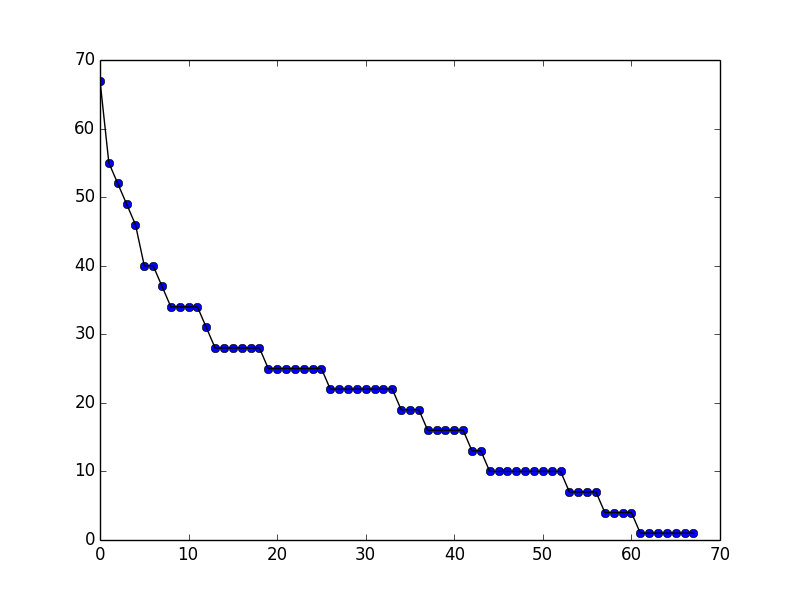
Voici les résultats d'une simulation du modèle de l'urne avec les paramètres : 5 boules initiales +3 buzz +1 innovation. La première figure représente le nombre de couleur d'une certaine couleur en fonction de son ordre d'apparition dans l'urne. Dans la deuxième les valeurs sont ordonnées par nombre de boules.
L'allure de la courbe est influencé par les paramètres mais on peut s'attendre à ce genre de courbe pour des données obéissant à une loi de Zipf.
Dans nos simulations, nous avons pris un article sur Pubmed (ou plusieurs traitant un même thème) et extrait ses mots clés. On se place alors dans l’état initial de l’urne où les différents mots correspondent à des boules de différentes couleurs. Ensuite, nous avons regardé les mots-clés des articles citant celui de départ. On regarde un par un les mots-clés d’un article. Si le mot-clé est déjà présent dans l’urne, on augmente le nombre de boule de sa couleur. Sinon, on introduit une boule d’une nouvelle couleur (i.e. on ajoute ce mot dans l’urne). Ce mot correspond à l’innovation.
On répète le processus avec les articles qui ont cité l'article initial. On avance ainsi dans le temps.
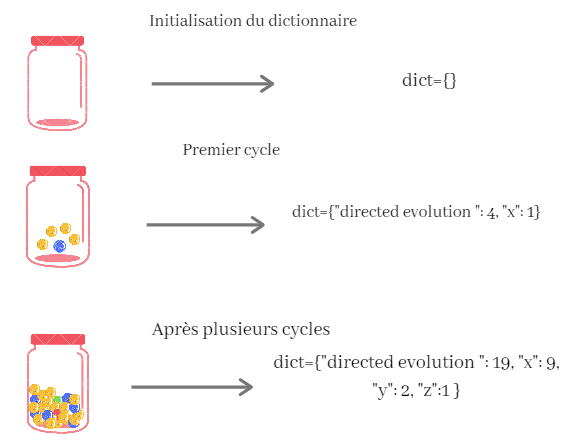
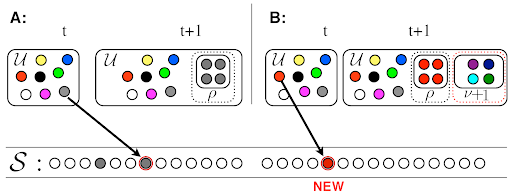
Dans l’algorithme de notre outil, l’urne sera remplacée par un dictionnaire, dont les clés sont les termes apparaissant pendant l’étude et qui représentent les boules de couleurs. La valeur associée à ces clés correspondra au nombre de fois où le terme apparaît pendant l’étude, que l’on peut apparenter au nombre de boules de même couleurs dans l’urne. Grâce au package Entrez de Biopython, on lance la recherche du thème sur Pubmed et on récupère un dictionnaire.
Nous avons dû choisir des thèmes dont la première recherche ne donnait pas un nombre de résultats trop grand. En effet, pour chaque article, nous récupérons ceux dans lesquels ils sont cités. Nous avons choisis les thèmes suivants :
Les thèmes sont composés d'un ou de plusieurs mots. Ce détail a été difficile à gérer dans la conception de notre code.
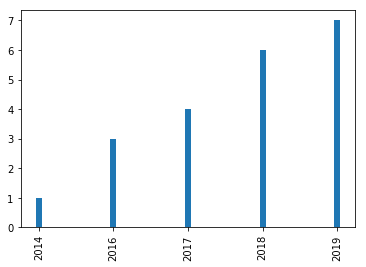
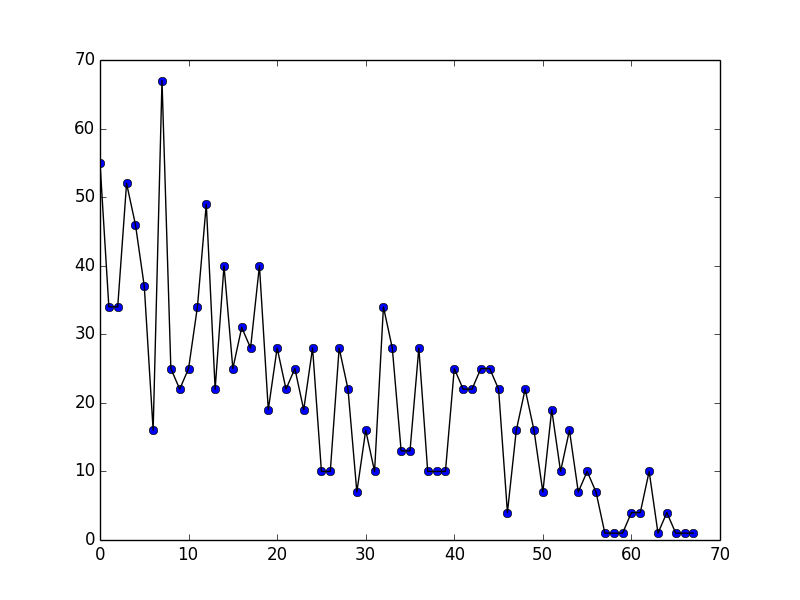
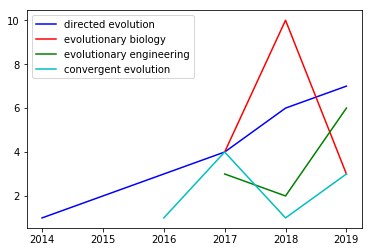
Le graphique ci-contre présente le nombre d'occurences du terme "directed evolution" en fonction de l'année. Pour ce cas, nous n'avons pas donné d'article initial. Lors de l'exécution du code, la libraire Biopython lance une recherche sur le terme "directed evolution" et prend comme article initial celui qu'elle considère comme le plus intéressant. Aux vues de notre graphique, il ne faut donc pas conclure que la première occurence de "directed evolution" date de 2014. L'année 2014 est en réalité l'année de publication de l'article que Biopython a pris en référence. Cependant, nous pouvons noter une augmentation de l'apparition du terme après cette date.
Ces résultats sont cependant peu fiables. En effet, il faudrait réaliser un plus grand nombre d'itérations et effectuer l'étude sur un plus grand nombre d'années pour voir apparaître une tendance et conclure plus certainement.
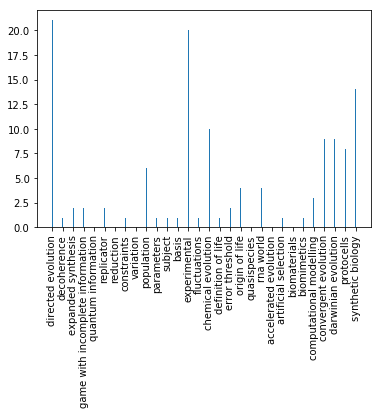
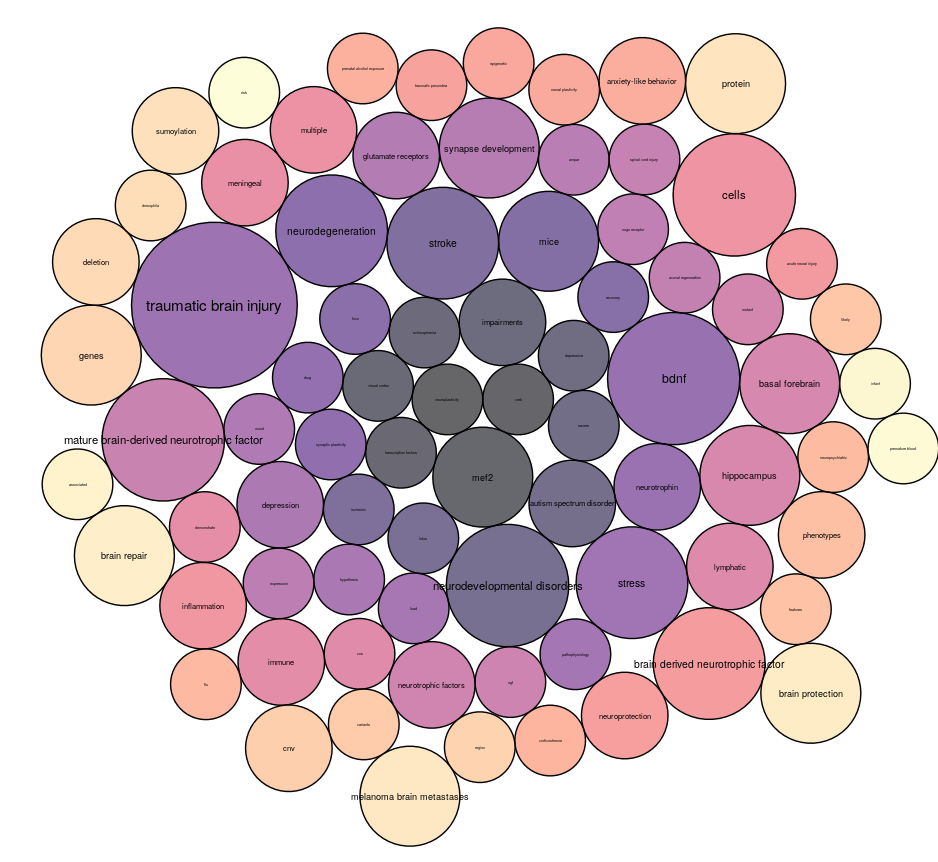
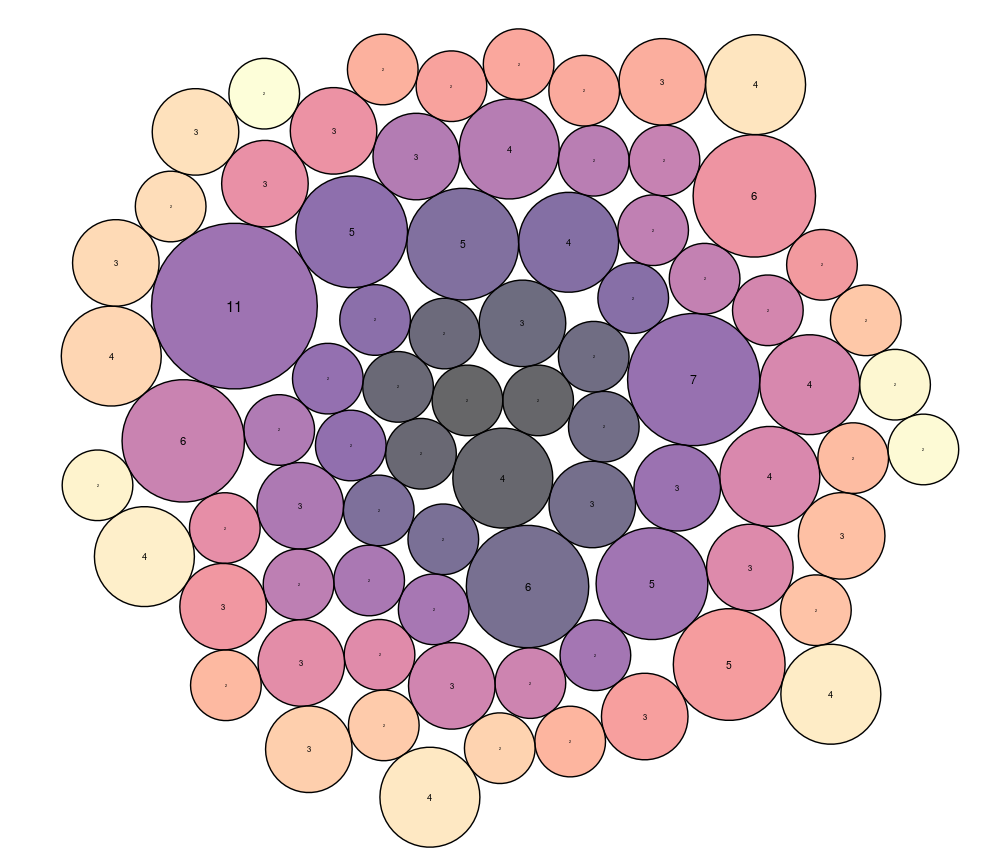
Le graphique ci-contre présente une partie des termes obtenus après exécution de notre code avec l'exemple "directed evolution". Beaucoup de mots ont été obtenus, nous n'en avons représenté que quelques uns, dans l'ordre d'apparition à partir de l'article initial.
Nous pouvons noter que les mots obtenus ont des nombres d'occurence très différents les uns des autres. Nous obtenons certains résultats intéressants, comme les nombreuses apparitions des termes :
Nous obtenons également de nombreuses fois les termes "population" ou "experimental". Il faut bien sûr ne pas les considérer car ce sont des moments très courants dans le vocabulaire scientifique. Ils ne traduisent donc en rien un phénomène d'innovation.
Pour extraire nos données, pour chaque thème, nous avons initialisé une urne avec 10 articles. Par la suite, nous avons réalisé 5 cycles pendant lesquels on prélevait 5 articles citant les articles précédents. 2 types de représentation pour les résultats:
Représentation 1: Courbes avec dates
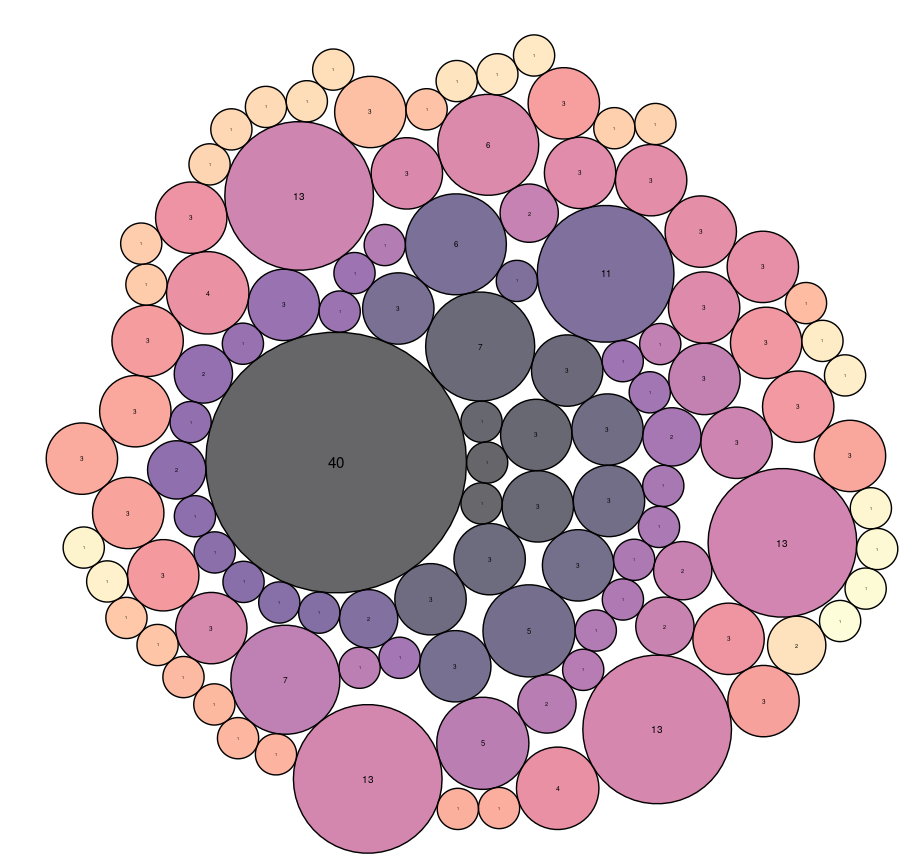
Représentation 2: Représentation boule via R
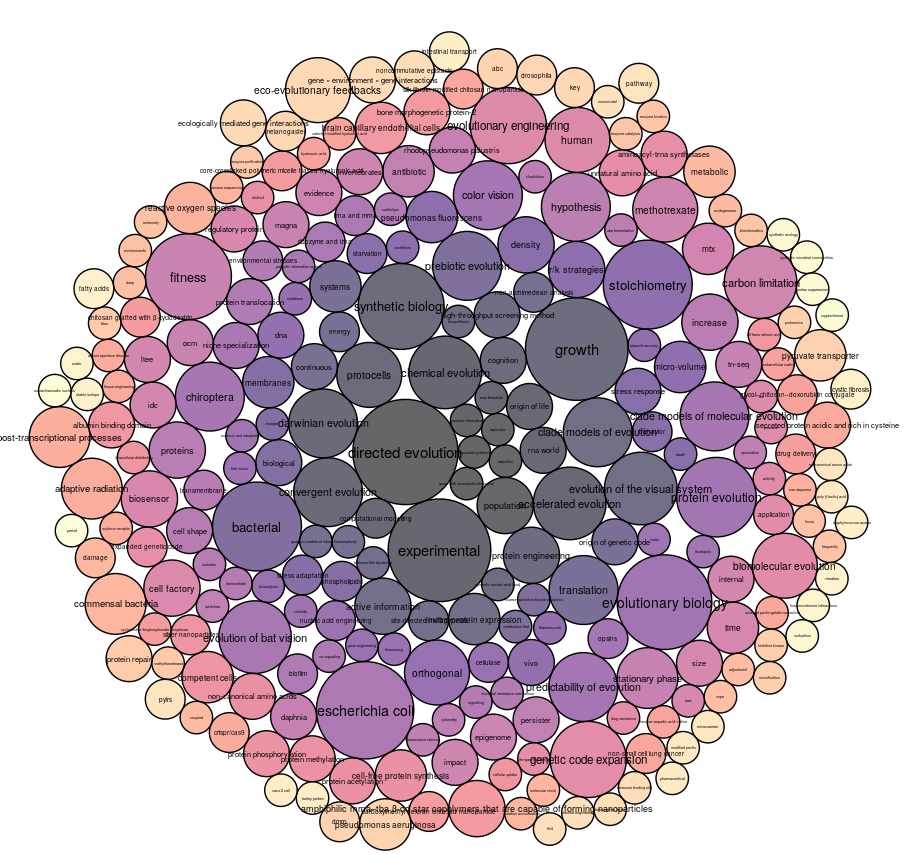
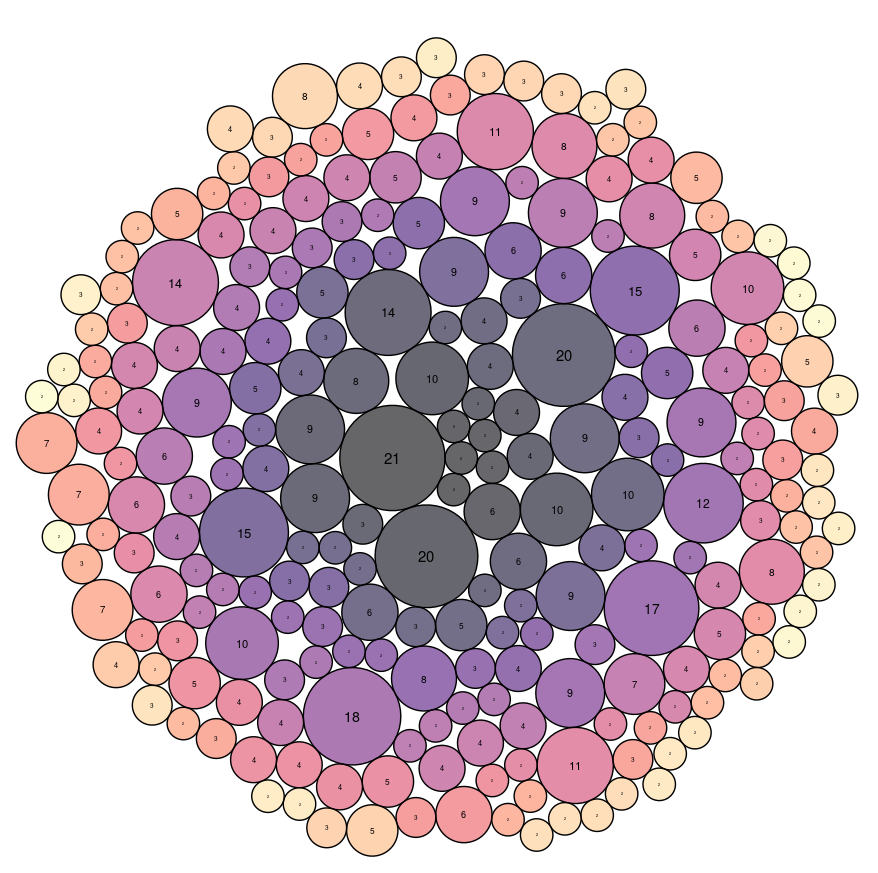
Nous remarquons que la boule la plus importante correspond au thème initial ("directed evolution"), ce qui est cohérent. Les articles citant l'article de départ ont de grandes chances de traiter le même thème.
Les autres thèmes très représentés sont "growth", "experimental", "synthetic biology", "stoichiometry", "fitness", "echerichia coli", "darwinian evolution", "convergent evolution" et "evolutionary engineering".
On retrouve alors des termes fréquents en biologie comme "echerichia coli", "growth", "experimental" et "fitness". Ils ne sont pas très pertinents pour notre étude.
Certains sont plus intéressants comme : "darwinian evolution", "convergent evolution" et "evolutionary engineering". Ceux-ci semblent plus spécifiques. Nous pouvons donc nous demander si le thème "directed evolution" est un précurseur de ces trois thèmes, ou un dérivé. Il faut alors analyser les dates d’apparition pour en savoir plus.
Evolutionary engeneering: C’est une méthode alternative de synthèse des biopolymères avec des fonctions et des propriétés désirées particulières en utilisant avec avantages les principes de l’évolution naturelle.
Convergent evolution: dans la biologie de l’évolution, convergent evolution désigne le fait que des organismes qui ne sont pas étroitement liés (non monophylétiques), développent indépendamment des traits similaires du fait qu'ils doivent s'adapter à des environnements ou des niches écologiques similaires. (opposé d'évolution divergente)
On pourrait changer l’algorithme et plutôt partir d’un article + un thème au lieu d’un thème, nous avons tenté ce changement avec le thème précédent "directed evolution":
Le graphe ci-dessus représente le nombre d'articles par année contenant les termes liés à "directed evolution" dans notre jeu de données. Les termes liés apparaissent après mais cela n'apporte guère d'informations sur les relations entre les thèmes. D'une part, le nombre d'articles par année n'est pas le même. Par ailleurs, la première apparition dans notre jeu de données ne reflète pas la première apparition dans le domaine de la biologie. Les courbes sur le graphe ne permettent donc pas de conclure à un lien entre les différents termes obtenus. Nous ne pouvons pas dire qu'un thème découle d'un autre mais seulement que les deux sont liés ou qu'un a été utilisé pour l'étude d'un autre.
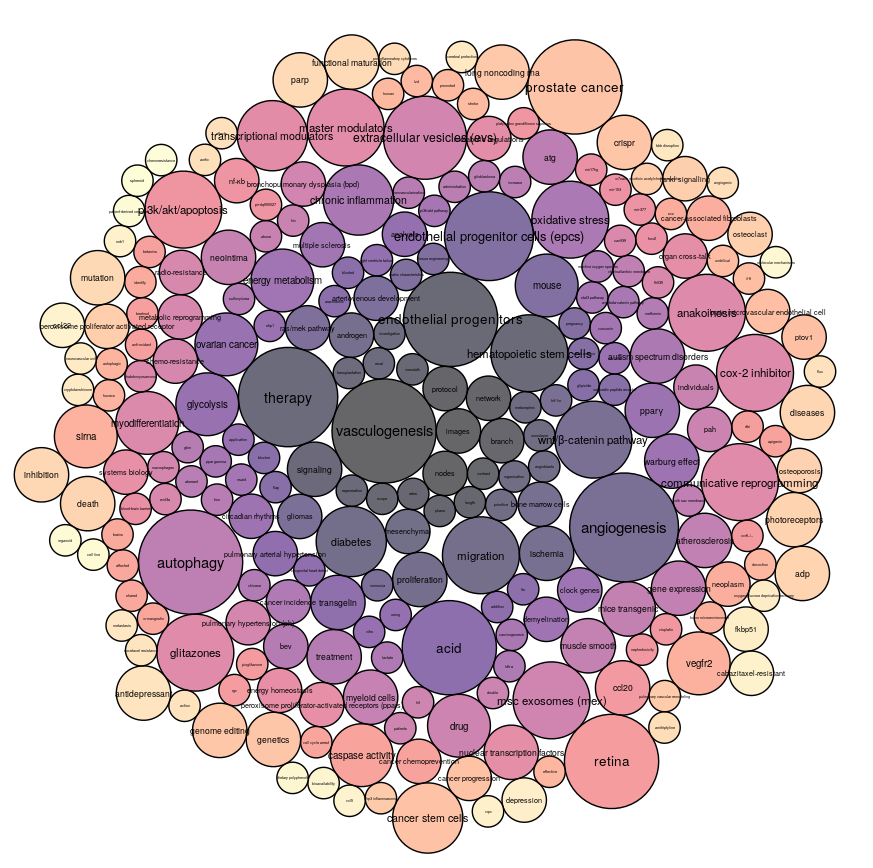
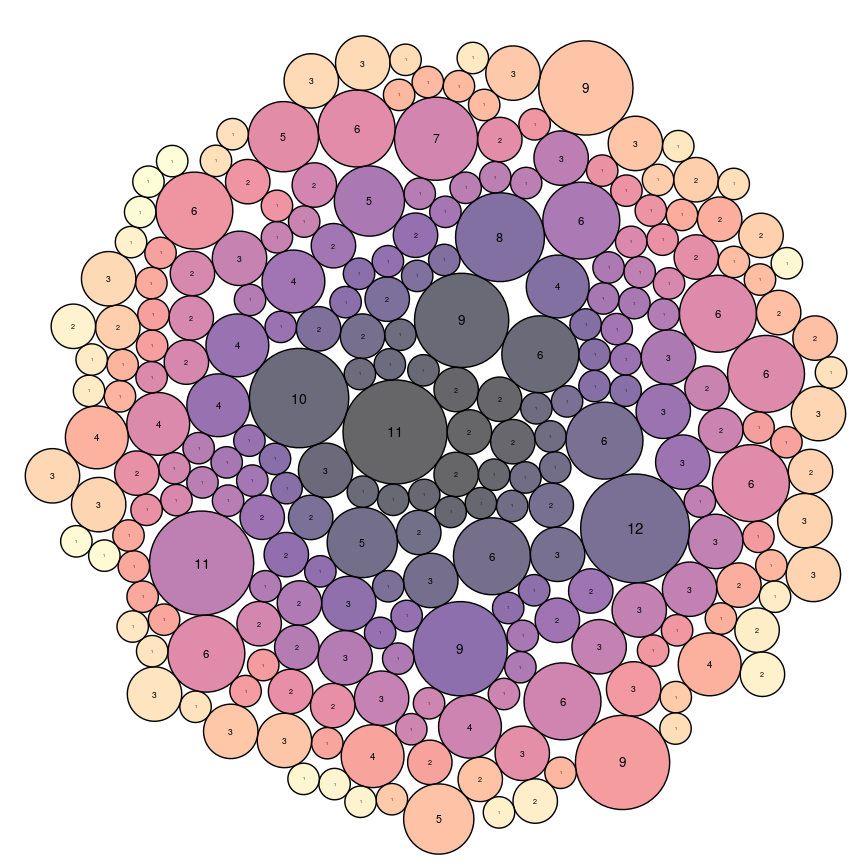
A la fin du processus, l’aire du terme "vasculogenesis" n’est pas la plus importante, c’est un autre terme proche "angiogenesis" qui présente l’aire la plus importante, il serait donc intéressant d'étudier ce terme. En effet, la vasculogénèse concerne l’étude de la formation des premiers vaisseaux sanguins et lymphatiques tandis que l'angiogénèse concerne la création de vaisseaux à partir de vaisseaux sanguins existants.
Pour le graphique ci dessous (avec dates), nous avons réunis les termes "endothelial progenitors" et "endothelial progenitor cells"
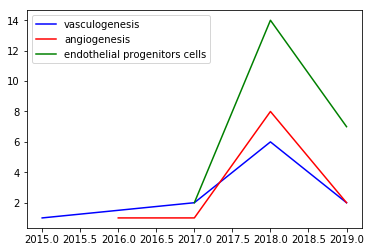
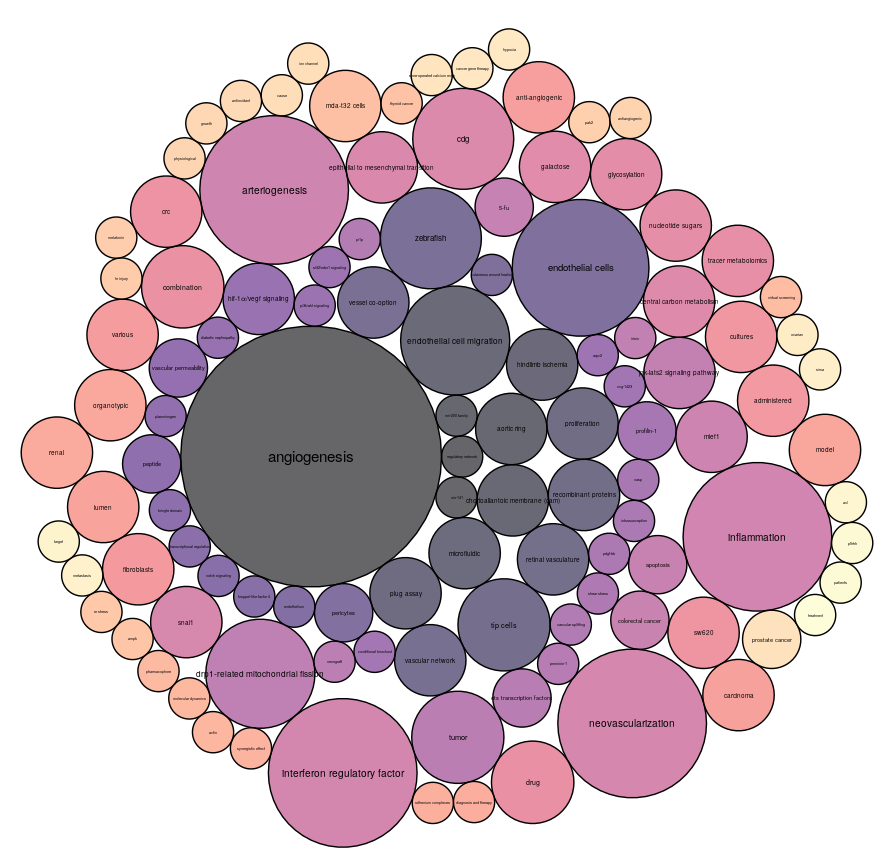
En observant son évolution via l’outil, on peut voir que l’aire du terme "angiogenesis" est largement supérieure aux autres.
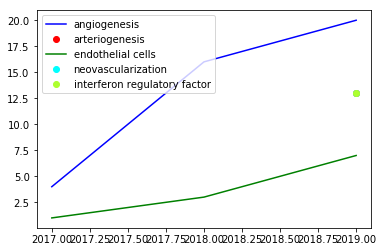
L'artériogénèse est la formation spécifique des artères. L'endothélium vasculaire est la couche la plus interne des vaisseaux sanguins. Les termes obtenus sont donc cohérents car sont très liés au thème initial.
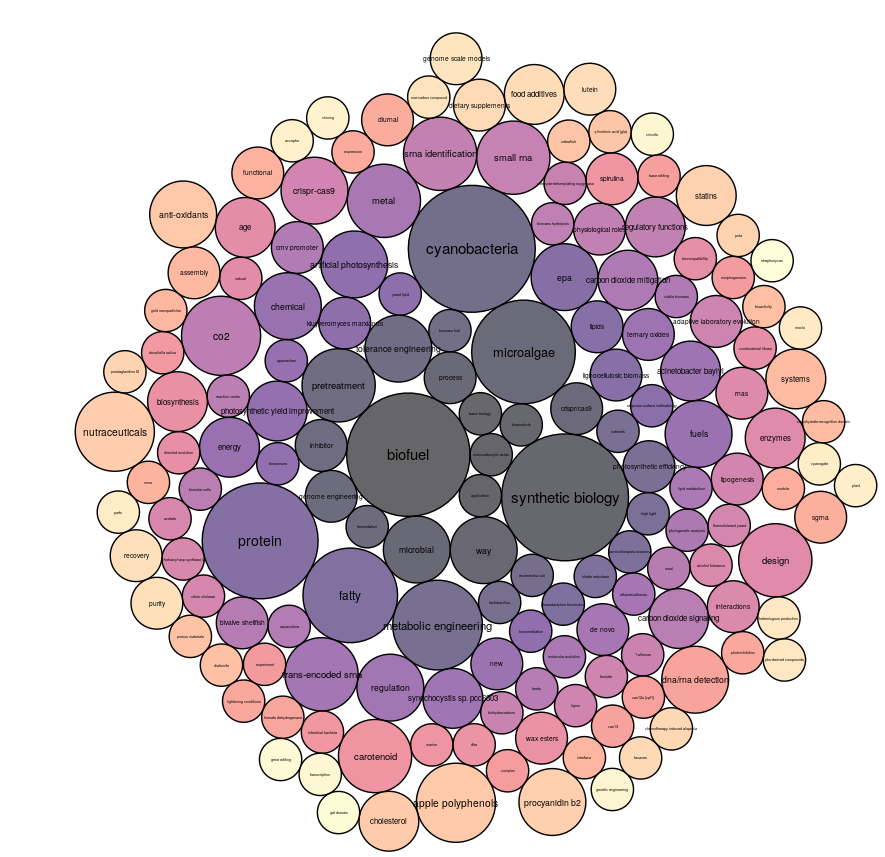
Les résultats pour biofuel sont assez intéressants, on retrouve des termes en relation avec le domaine d’étude des carburants biologiques. On retrouve notamment les termes "cyanobacteria" et "synthetic biology" que l’on pourrait plutôt assimilé à des termes en relation avec "biofuel" que des termes innovants. On peut tout de même comparer l’évolution des ces thèmes au cours du temps.
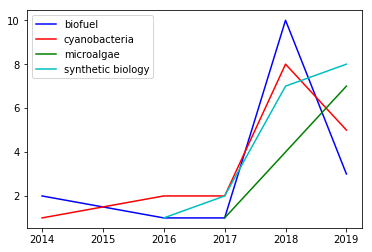
Dans le graphe avec les dates, il est intéressant de noter que les courbes des termes retenus ont une allure semblable. Nous pouvons penser que ces termes apparaissent dans les mêmes articles. Nous ne pouvons pas conclure quant à l'innovation mais conclure que ces termes sont très reliés.
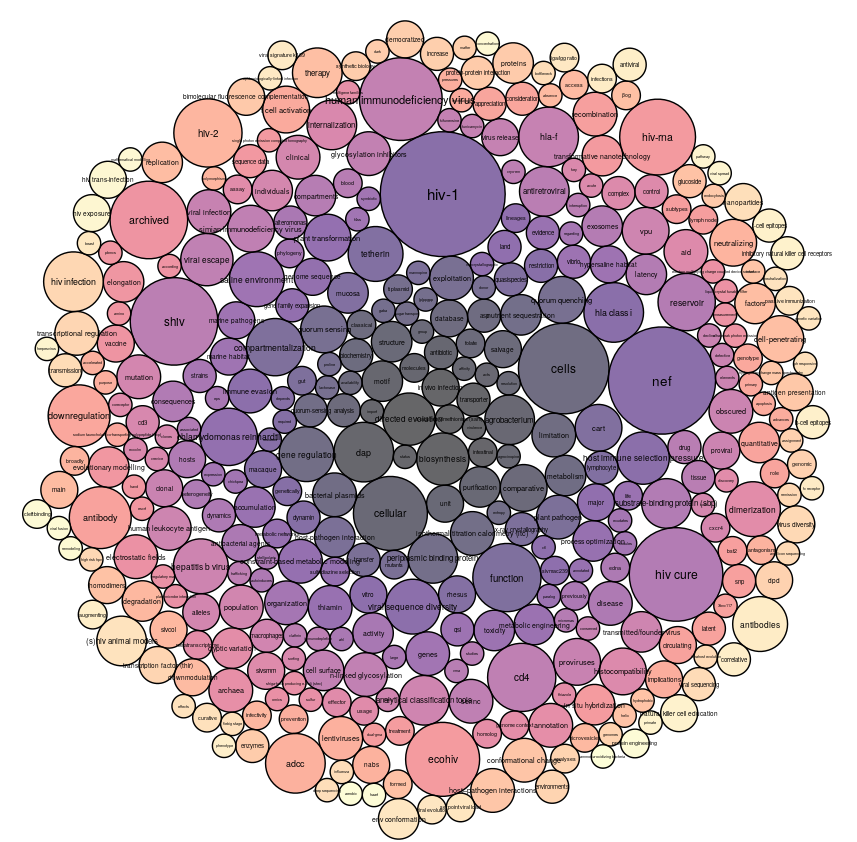
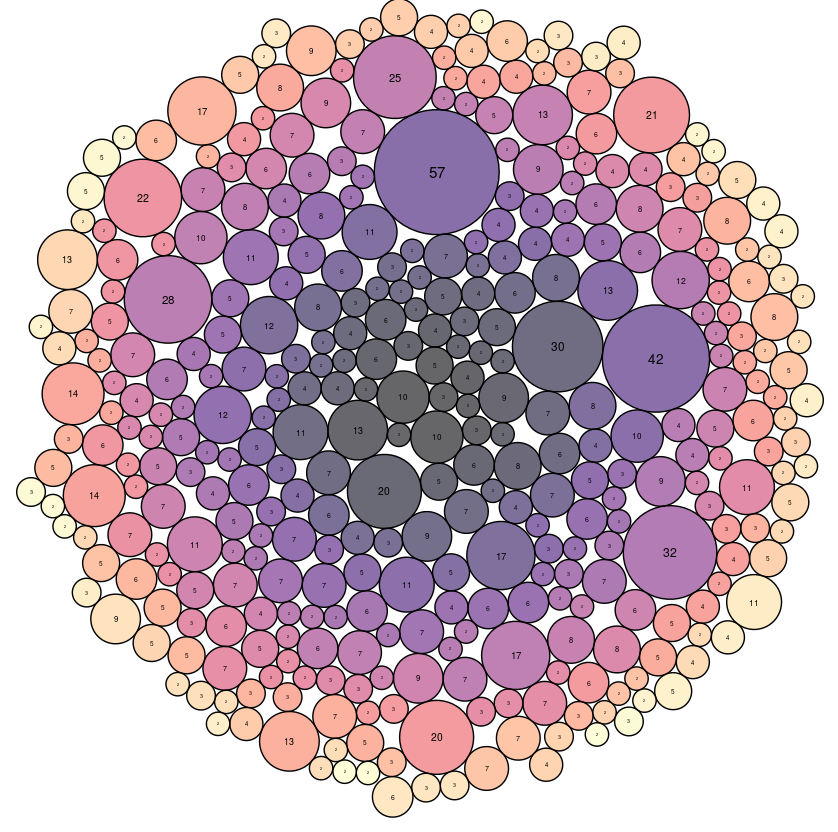
Nous pouvons observer que cette méthode est moins pertinente pour analyser les termes liés (ou de l’innovation reliée à un thème), puisqu’on observe qu’on dérive rapidement dans un autre sujet (dans ce cas HIV).
Cependant, cette méthode permet d'observer les termes dérivés de notre article de base.
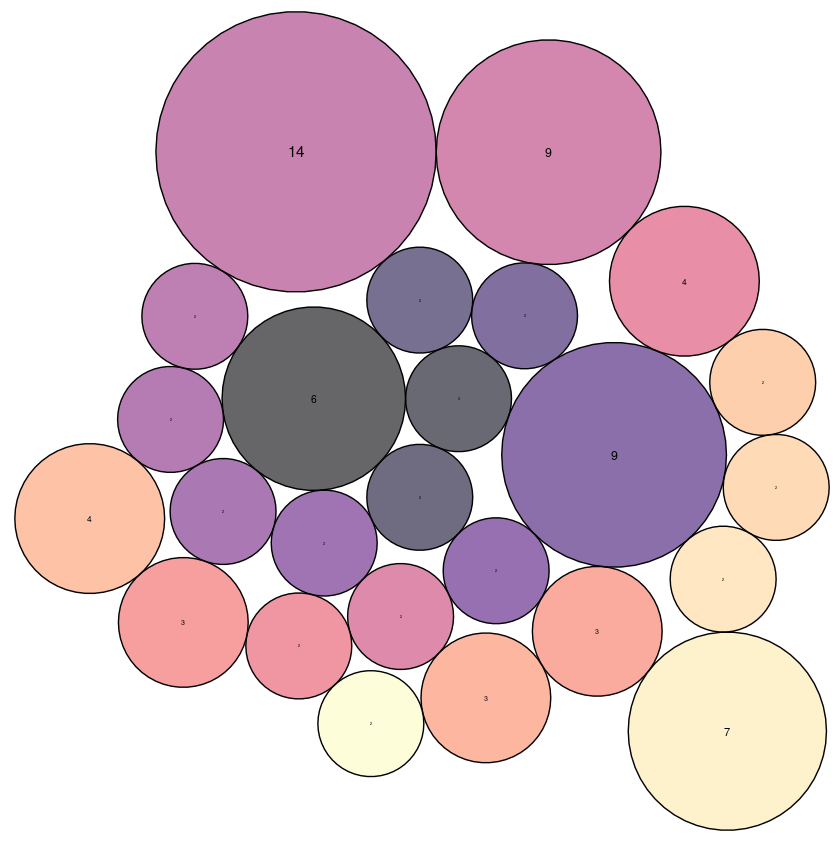
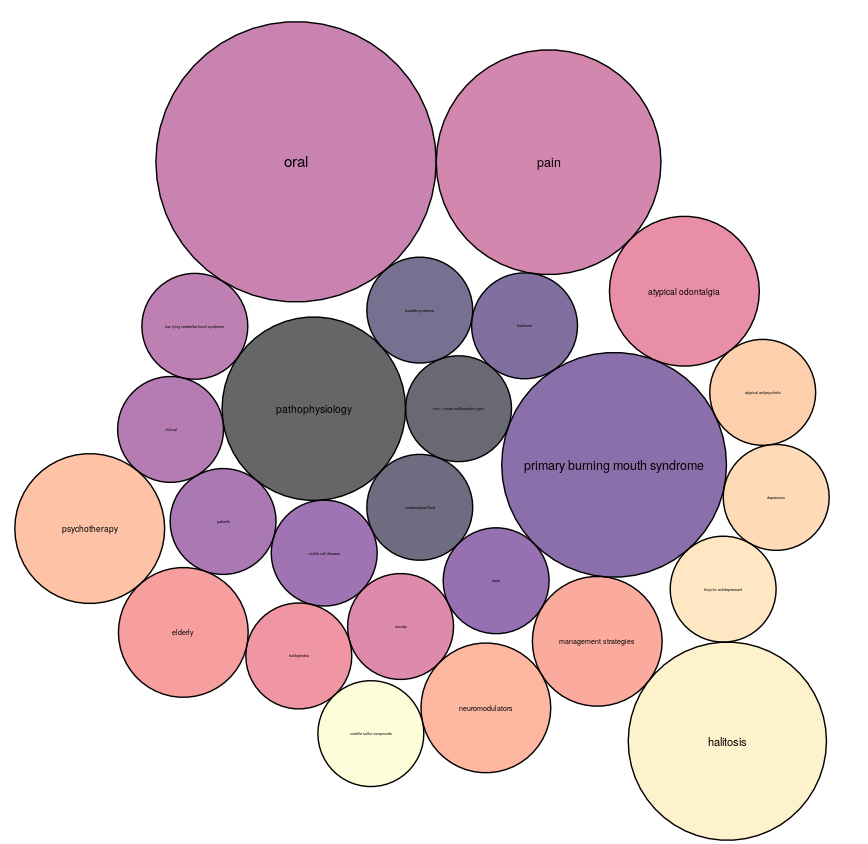
Il est important de remarquer que les graphiques ci-dessus nous indiquent surtout les termes/articles corrélés et non pas de l'innovation. Cela peut tout de mếme être intéressant car l’outil nous amène alors à découvrir d’autres termes reliés pouvant nous aider dans nos recherches.
Malgré la forte apparition des termes ci-dessus, il est difficile ici de savoir s'il s'agit de termes qui sont apparus avec "directed evolution", ou après ou s'ils existaient déjà avant. La façon dont nous avons réalisé notre étude ne nous permet pas de conclure à un caractère innovant ou non. Il faudrait pour cela étudier la date de première apparition de ces thèmes, et voir s'ils étaient dès le début liés à "directed evolution". Nous pourrions alors conclure que le terme est né suite à l'étude de "directed evolution".
L'innovation ne passe pas nécessairement par la création de nouveaux mots mais également par l'association de termes existants. Par exemple, les cyanobactéries peuvent avoir été découvertes il y a assez longtemps, mais leur étude en rapport aux biofuels peut être quelque chose de récent (et donc à caractère innovant). Il s'avère aussi donc intéressant d'étudier la co-occurence de deux termes au sein d'un article.
Notre objectif principal dans la réalisation de ce projet était de produire un programme permettant de détecter l’innovation et le développement d’un thème en particulier dans la littérature biologique. Malheureusement, les résultats obtenus ne permettent pas vraiment de caractériser l’innovation mais plutôt de trouver les thèmes reliés ou dérivés du thème initial.
En effet, la plupart des thèmes que nous avons obtenus à partir des thèmes initiaux sont pertinents. Il faudrait encore trier les termes obtenus pour supprimer les termes généraux du vocabulaire biologique (“growth”, “population”, ...). Cela nous indique ainsi que le modèle de l’urne que nous avons utilisé est plus ou moins respecté par les données. On obtient des termes liés au terme de départ avec des fréquences élevées. L’apparition de nouveaux mots semble aussi engendrer l'apparition d’autres nouveaux mots. La difficulté réside tout de même sur le fait de déterminer si l’apparition d’un mot correspond à de l’innovation dans la réalité.
Ce projet nous a également permis de découvrir d’autres fonctionnalités pour notre outil. En effet, nous avons observé qu’en partant d’un thème et d’un article de référence, il était possible de voir apparaître des thèmes très divers, selon les articles choisis par Pubmed.
Aux vues des résultats que nous obtenons, notre outil pourrait permettre aux chercheurs d’orienter leurs recherches bibliographiques. En rentrant un thème sur lequel ils veulent s’orienter, notre programme leur rendrait les thèmes liés au thème initial. Notre outil leur permettrait également d’analyser l’influence de ses recherches sur la communauté scientifique.
Durant notre projet, nous avons dû faire face à de nombreuses difficultés. Dans un premier temps, il est évident que l’algorithme présente des résultats plus efficaces lorsque le nombre d’itérations est assez grands. Cependant, dans de nombreux cas la quantité de données à gérer était beaucoup trop importante pour les ordinateurs utilisés. Nous avons ainsi dû restreindre certains paramètres de notre algorithme. De plus, les moteurs de recherche tels que PubMed sont assez contrôlés et bloquent parfois l’accès aux informations quand le nombre de requêtes est conséquent. Enfin, réaliser un algorithme basé sur l’analyse de texte a également été assez fastidieux car il nous a fallu traiter cas par cas chaque exceptions avec précision.
Nous tenons à remercier notre tuteur de projet Sergio PEIGNIER. Ses conseils nous ont permis d'orienter nos recherches sur un thème original et peu documenté dans la bibliographie. Son apport de codes Python a également été une précieuse aide.
Merci également à Carole KNIBBE et Nicolas PARISOT pour leurs conseils à mi-parcours de ce projet.
N'hésitez pas à nous contacter pour tout complément d'informations sur ce projet.

Responsable traitement des données
sarah.dandou@insa-lyon.fr

Responsable récupération des données
catalina.gonzalez-gomez@insa-lyon.fr

Responsable modèle théorique de l'urne
luka.matsuda@insa-lyon.fr

Responsable site web
maxime.vincent@insa-lyon.fr